elemeket
absztrakt
Az elmúlt 30 évben számos alapvizsgálatot végeztünk két Oryza sativa subsp. indica, Zhenshan 97 (ZS97) és Minghui 63 (MH63). Ezen tanulmányok felbontásának javítása érdekében két referencia-genom-összeállítást hoztunk létre a legkorszerűbb szekvenálási technológiák felhasználásával. A PacBio SMRT technológia használatával több mint 108 (ZS97) és 174 (MH63) Gb nyers szekvencia adatot állítottunk elő 166 (ZS97) és 209 (MH63) BAC klóncsoportból, és generáltunk
74 (MH63) Gb teljes genom párosított végszekvencia (WGS) szekvencia adatai Illumina szekvenálási technológiával. Ezekkel az adatokkal sikeresen összeállítottunk két közzétett platina szabványt. Itt találhatók a nyers adatok teljes készletei, amelyeket e két referencia genomi együttes előállításához használtak. Ezek az adatkészletek felhasználhatók új programok tesztelésére a jobb genom-összeállítás és annotáció érdekében, segíthetnek új perspektívák felfedezésében a genom felépítésével, működésével és evolúciójával kapcsolatban, valamint elősegíthetik a szükséges támogatást a biológiai kutatáshoz általában.
Metaadatok összefoglalása
Töltse le a metaadatfájlt
Gép által elérhető metaadatfájl, amely leírja az adatokat (ISA kártya formátum)
Háttér és összefoglalás
mód
BAC könyvtár építése és szekvenciális szekvenálás
A tanulmányban használt két BAC könyvtárat korábban közzétették 11. Röviden, az egyes fajtákból részben emésztett (azaz Hin dIII) és méret szerint szelektált genomi DNS-t klónoztunk a pAGIBAC1 Hin dIII helyére, és transzformáltuk kompetens Escherichia coli DH10B T1 sejtekbe. Mindkét OSIZBa (ZS97) és OSIABa (MH63) nevű könyvtár 36 864 BAC klónt tartalmazott, az átlagos inzertméretek
125 kb (MH63) és bevonattal láttuk el
10,7 × (MH63) az egyes genomokból 11. Ezenkívül 33, 969 (ZS97) és 35, 549 (MH63) kétirányú BAC végszekvenciát (BES) generáltunk az egyes könyvtárak első felében 11.
Fizikai térképek
Az SNaPshot ujjlenyomat-módszerrel fent leírt alacsony lefedettségű (PM) fizikai térképeket fentebb leírtuk11. A két PM-t a KeyGene Whole Genome Profiling (WGP) módszerrel 12 alakítottuk át. A WGP FingerPrint Contig (FPC) PM-eket négy lépésben állítottuk össze: (1) BAC DNS előállítása, (2) WGP BAC plazmidok előállítása adapterekkel indexeléshez és szekvenáláshoz, (3) Illumina szekvenálás és (4) bioinformatikai feldolgozás. A 4. lépésben WGP dekódoló szkriptek alkalmazásával 99 996 (ZS97) és 103 609 (MH63) egyedi WGP címkéket oldottunk fel, és 32 829 (89,1%) és 30 749 (89,3%) BAC-t jelöltek meg a ZS97 és MH63 könyvtárakban, ill. WGP szekvencia címkéket használva minden könyvtár BAC klónjához, két új PM-t készítettünk az FPC 13 szoftverrel (9.4 verzió). Manuális szerkesztés és a korábbi SNaPshot PM-ekkel történő integrálás után a továbbfejlesztett ZS97 és MH63 PM-k 539 és 401 folytatásból álltak, amelyek 28, 372 és 24, 519 klónokat, valamint 4 457 és 6230 klónokat jelentettek szingulettként. A teljes méretek nagyságát 342 Mb-ra becsülték a ZS97 esetében (N50 = 940 kb) és 349 Mb-ot MH63 esetén (N50 = 1270 kb).
A PacBio BAC klón szekvenálása
Az egyes PM-ek minimális útburkolási útvonalának (MTP) BAC klónjait automatikusan kiválasztottuk egy testreszabott szkript használatával, és kézzel OSZBzz (ZS97) és OSIABzz (MH63) feliratokkal ellátott MTP könyvtárba rendeztük és -80 ° C-on tároltuk. 4751 BAC MTP-klónt gyűjtöttünk össze a ZS97 és MH63 esetében. Az MTP klónok teljes listája az 1a-b. Kiegészítő táblázatban található.
A PacBio BAC klón szekvenálásához az MTP BAC klónokat oltottuk 96 lyukú mély lyukakba, egy éjszakán át 37 ° C-on növesztettük, centrifugáltuk a sejtek pelletálása céljából, majd felhasználásig -80 ° C-on tároltuk. Ezután a BAC-kat úgy alakították ki, hogy a fagyasztott blokkokból származó üregeket összevonták a hat konfiguráció egyikébe: azaz egyhajós társulások (12 BAC-k medencénként), két soros asszociációk (24 BAC-k medencénként), négy oszlop-csoportok (32 BAC-k medencénként), 6 csoportos oszlopok (48 BAC készletenként), nyolc oszlop társítás (64 BAC készletenként) vagy teljes ellátás csoportok (96 BAC készletenként). Ezután mindegyik keverékből DNS-t extraháltunk egy standard alkalikus lízis plazmid DNS 14 plazmid izolációs protokoll alkalmazásával. Összesen 166 (ZS97) és 209 (MH63) készletet szekvenáltunk (lásd részletes pooling sémánkat a 2a-b. Kiegészítő táblázatban). 16 μg összegyűjtött plazmid DNS felhasználásával PacBio szekvencia könyvtárakat készítettünk a következő gyártói protokollok szerint, a 20 kb méretű Blue Pippin templát előállításához leírva. Az SMRT szekvenálást PacBio RSII eszközön végeztük P5/C3 szekvencia kémia és 3 órás filmek felhasználásával.
Nyers olvasási produkció a PacBio-val
A ZS97 és az MH63 BAC szekvenciák mindkét csoportjának alláncelemzését a PacBio SMRT Portal (2.3.0 verzió) segítségével végeztük el. A ZS97 esetében 227 SMRT sejtből származó adatokat (amelyek az újrareakciókat számlálják) elválasztották és szűrték (azaz az RS_Subreads protokoll használatával a minimális polimeráz leolvasási hossza = 50 bp, a minimális polimeráz leolvasási minősége = 75 és a minimális alrész hossza = 50 bp), ami összesen 107,5 Gb felhasználható szekvenciaadat eredménye (a polimerázreakciók teljes száma = 11,6 M, a polimerázszám N50 = 12,8 kb; a merülések teljes száma = 17,7 M, a szubkután átlagos hossza = 5,7 kb, az alsósor N50 = 8,0 kb). Az MH63 esetében 317 SMRT sejt adatait dolgoztuk fel a fentiek szerint (174 Gb felhasználható adat; 18,2 M polimeráz leolvasása, a polimeráz N50 = 12,1 kb leolvasása; 26,8 M részláncok, átlagos alrész hossza = 5,5 kb, alrész N50 = 7, 8 kb).
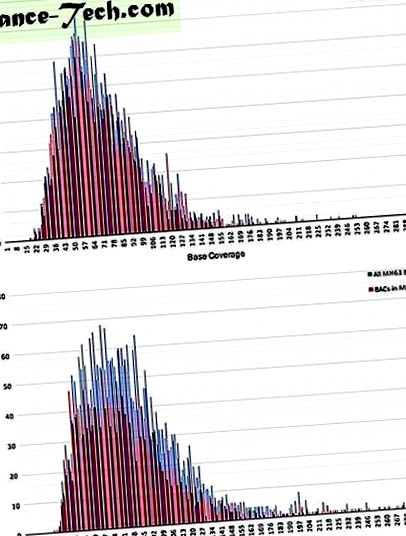
PacBio adat összeállítás és BAC szekvencia azonosítás
Teljes méretű kép
Növényi anyag, DNS könyvtár építése illumin szekvenáláshoz
Az Illumin rövid leolvasási szekvenálási technológiáját is alkalmaztuk a ZS97 és MH63 genomok szekvenálására, a teljes genom-puskás (WGS) megközelítést alkalmazva. Növényi anyagokat üvegházban növesztettünk, és 4 hetes leveleket használtunk a genomi DNS kinyerésére standard eljárások alkalmazásával. Párvégű könyvtárak, beleértve a kis betéteket (
300 bp) és két nagy betétkönyvtárat (5 kb, 10 kb) készítettünk Illumina párosított vég- és mat-pár készletekkel (1. táblázat). Legalább 5 μg genomi DNS-t töredezett nitrogéngázzal történő porlasztással fragmentáltunk a rövid inszert beillesztésű végkönyvtárakhoz. Nagyobb mennyiségű, jó minőségű genomi DNS-re (10-30 μg) volt szükség egy hosszú mat-pár inzertet tartalmazó könyvtár felépítéséhez. Az Illumina szekvencia könyvtárakat a gyártó protokollja szerint készítettük el. A könyvtárakat az Illumina HiSeq 2000-n szekvenáltuk a szokásos Ilulina-protokollok szerint (Illumina, San Diego, Kalifornia). Az egyes fajtákra generált nyers szekvenciaadatok teljes mennyisége
97, 5 Gb adat a ZS97 és
74,0 Gb adat az MH63-hoz. Adatszűrési lépések sora után a PCR duplikáció és az adapter szennyeződése által okozott mesterséges leolvasások eltávolítására használták őket, összesen - 87, 4 Gb magas minőségű (> 200 ×) olvasmányt a ZS97 és
67,9 Gb (> 170x) MH63-hoz jutunk (1. táblázat). A könyvtár minőségét a betétméretek megoszlásának és a szekvencia mélységének meghatározásával ellenőriztük. A tényleges betéthosszakat a párosított végek O. sativa subsp. japonica cv. Nipponbare referenciagenom (Nipponbare RefSeq) 17 .
Asztal teljes méretben
Az Illumina az előfeldolgozást és a de novo összeszerelést olvassa
Egy új hibrid megközelítést alkalmaztunk, amely ötvözte a de novo összeszerelés és a referencia módszereket 18 az Illumina-leolvasások összeállításához az egyes genomokhoz. A ZS97 és az MH63 összes szekvenált leolvasását korrigáltuk a Medusa 19-gyel és a Quake 20-mal. A javított leolvasásokat a végén csonkoltuk, ha a szekvencia minőségük kevesebb mint 20 volt a fastx_tool_kit (//hannonlab.cshl.edu/fastx_toolkit/index.html) használatával, és a számlálókat eltávolítottuk a cutadpat 21 (//github.com/) Marcely segítségével/cutadapt /). A feldolgozott adatokat ezután BWA 22 segítségével leképeztük egy Nipponbare RefSeq-re. Minden folytonos területre leképezett leolvasást egyenként vettünk, és az összefüggő lefedettségi területet blokkként határoztuk meg. A blokkok és a szuperblokkok meghatározása megegyezik a fentebb leírtakkal 23, azzal a különbséggel, hogy a minimális szuperblokk-hosszunk 20 kb, a szuperblokk-átfedés pedig 2 kb volt. Helyileg de novo összeállítottuk a szuperblokkban összegyűjtött összes olvasatot a SOAPdenovo 24 segítségével. Különböző k-merek sorozatát teszteltük, és a fennmaradó, a legmagasabb N50-értékeket tartalmazó sejteket megtartottuk. A kapott folytatásokat az AMOS 25-gyel állítottuk össze referencia-kromoszómáik felhasználásával. A leképezés nélküli leolvasásokat feltérképeztük az indica 9311 genomra, és ugyanazon eljárással állítottuk össze.
Ezután a Nucmer 27-et használták az összes szupervezérlés összehangolásához a Nipponbare RefSeq-hez. Ezután ellenőriztük a lefedettség mélységét és a szomszédos területek közötti hézagok feltérképezését. A leképezett leolvasásokat úgy választottuk ki, hogy az MH63 és ZS97 genomokban áthidalott réseket meghosszabbítottuk 200 bázissal mindkét rés mindkét oldalán. Az összes kiválasztott számot ezeken a területeken a cap3 28 segítségével állítottuk össze. Az összeállított kontigákat két folyamatos szupervezérléssel igazítottuk, és egymás utáni elrendezésük alapján kapcsoltuk össze.
A Nipponbare és a 9311 genomokban nem szereplő specifikus MH63 és ZS97 szekvenciák beépítéséhez teljes de novo genom összeállítást hajtottunk végre az összes feldolgozott SOAPden 24 leolvasás felhasználásával, majd összehangoltuk a kombinált szupravezetőkkel összeállított de novo állványokat, és tovább ligáltuk a megfelelő szupervezérléseket. Végül az állványokat az SSPACE 29 segítségével hajtották végre, és a réseket kitöltötték a gapCloser-rel (//sourceforge.net/projects/soapdenovo2/files/GapCloser/).
A WGS Illumina ZS97 és MH63 készletek végső statisztikáit a 2. és 3. táblázat tartalmazza, és a szomszédos PacBio kontigumok közötti hézagok kitöltésére használták.
Asztal teljes méretben
Asztal teljes méretben
Pszeudomolekulák építése két lépésben
Az első lépésben az összes BAC-szekvenciaadatot beillesztettük a Genome Puzzle Master 16-ba (GPM), hogy fizikai térképek és Nipponbare RefSeq 17 referencia kézikönyvek felhasználásával készítsünk PacBio-alapú szekvencia-összefüggéseket. A GPM egy félig automatizált csővezeték, amelyet arra fejlesztettek ki, hogy a logikai kapcsolati adatokat (pl. Fizikai térképeket) állványszekvenciákba integrálja kromoszómális tartományú szekvenciákba. Ennek eredményeként 318 (ZS97) és 216 (MH63) összeillesztett kontig van elrendezve és orientálva, valamint horgonyozva a megfelelő kromoszómájukra, kézi ellenőrzés, szerkesztés és a felesleges eltávolítás után. A PacBio-alapú végső összeállítások 3862 (ZS97, köztük 57 nem MTP) és 3254 (MH63, köztük 77 nem MTP) egyedi BAC szekvenciákból álltak.
Mivel ebben a tanulmányban fizikai térképalapú szekvenálási stratégiát alkalmaztunk, a WGP fizikai térképeink hiányosságai jelentik a PacBio-alapú összeállítások törésének fő okait. Ezért a második lépés a hiányosságok pótlása volt az Illumina WGS szerelési adatainak integrálásával. Azonban a potenciálisan helytelen adatok hatásának minimalizálása érdekében az Illumina adataiban csak a contra Illuminát használtuk, amely két szomszédos PacBio érintkezőt tudott teljesen összekapcsolni ebben a lépésben. A ZS97-ben összesen 81 hézagot (8 988 328 bp) és 35-et az MH63-ban (3 127 191 bp) töltöttünk 76 (ZS97) és 35 (MH63) Illumina folytatási szekvenciával (kiegészítő 4a-b. Táblázat). Ennek eredményeként a végső hibrid pszeudomolekulák (ZS97RS1 és MH63RS1) 237 (köztük 2 lezáratlan) és 181 (köztük 2 meg nem erősített) kontigént tartalmaztak a ZS97 és MH63 esetében.
Adatrekordok
Minden nem szekvenciális adat elérhető az iPlant Datastore-ban (4. táblázat). Az OSIZBa és az OSIABa BAC könyvtárak, vagy egyes BAC klónok beszerezhetők az AGI BAC/EST erőforrás központjában a www.genome.arizona.edu/orders címen. A BAC végszekvenciákat korábban a GenBankban tárolták KG737749 - KG771717 (ZS97, 1. adatcím: GenBank KG737749 - KG771717) és KG702200 - KG737748 (MH63, 2. adatszám: GenBank KG702200 - KG737748) hozzáférési számokkal.
Asztal teljes méretben
A nyers PacBio szekvenciaadatok elérhetők az NCBI Short Read Archive (SRA) SRP071597 (ZS97, 3. adatcím: NCBI Sequence Read Archive SRP071597) és SRP071598 (MH63, 4. Data Citation: NCBI Sequence Read Archive SRP071598) csatlakozási számok alatt. Az Illumina összes szekvenciaadata megtalálható az SRP071944 regisztrációs szám alatt (ZS97 és MH63, 5. adatcitáció: NCBI Sequence Read Archive SRP071944). Váratlan lemezhiba miatt elvesztettük a nyers PacBio szekvenciaadatokat 57 készletből. Szerencsére az összes HGAP futtatást archiválták az iPlant Datastore-ban a „halál-munkahelyek” alatt, ahol lehetőség nyílt e megfelelő csoportok szűrt alrétegeinek megszerzésére. Az Illumina által összegyűjtött adatok az NCBI közgyűlésen érhetők el GCA_001618795 (ZS97, 6. adatszám: NCBI assembly GCA_001618795) és GCA_001618785 (MH63, 7. adatszám: GCA_001618785) csatlakozási szám alatt.
Az egyes referenciagenomok végső genom-pszeudomolekuláit (1. verzió) az NCBI közgyűlésben GCA_001623345 (ZS97RS1, 8. adat hivatkozás: NCBI assembly GCA_001623345) és GCA_001623365 (MH63RS1, 9. adatszám: NCBI assembly_G0065) azonosítószámmal tároltuk.
Műszaki ellenőrzés
Lényegében mindegyik genomi ekvivalens BAC könyvtárat frissen növesztettük 384 lyukú lemezek lemásolt készleteiben, és háromdimenziós összevonást hajtottunk végre baktérium sejteken, majd sejtnövekedést és plazmid DNS extrakciót alkalikus lízis kémia alkalmazásával. A DNS-összeállításokat restrikciós enzimekkel (EcoRI/MseI) emésztettük, majd pool méretű oligomerek ligálását követtük, amelyeket úgy terveztünk, hogy specifikusan lokalizálják a BAC-klónok címét és asszociálódjanak a szekvenciákkal. A vegyes molekulák amplifikálása után Illumina szekvenálást hajtottunk végre, és a kapott adatokat elemeztük, hogy azonosítsunk egy 50 bp-os szekvencia taget a BAC-klón minden egyes címéhez, és sávkészleteket állítsunk elő az FPC-be bemenetként. Az FPC-t nagy szigorúságú beállításokkal (HS) futtattuk: először 'tolerancia = 0 [fix], levágás = 1e-15', majd a DQ kiválasztásával (3 lépésben: Levágás = 1e-18, 1e-21, 1e -24 ) a problémás folytatások terjesztésére használták. Az eredményül kapott HS PM-ek létrehozása után elvégeztük az Ends-to-end egyesítési lépést (Cutoff = 1e-9), és a címkézett szinguletteket beépítettük a kontigumokba (Cutoff = 1e-12), hogy korlátozott szigorúságú (RS) térképeket hozzunk létre. A WGP RS térképeket manuálisan módosítottuk, integrálva a korábbi alacsony PM-ek lefedettségével .
A BAC-alapú szekvenciaszekvenciák létrehozásának GPM 'assemblyRun' lépésében a két BAC-szekvencia összevonásának alapértelmezett paraméterei a következők voltak: 'minOverlapSeqToSeq = 1000 bp' és'identSeqToSeq = 99% ', és mindkét szekvencia végén átfedésekre volt szükség. A Nipponbare RefSeq 17-et használtuk referenciaként a kromoszómaszámok hozzárendeléséhez az összeszereléshez, valamint azok rendezéséhez és tájolásához. Ezenkívül a redundáns, egymást átfedő szekvencia csak egy példányát tartották meg az összeszerelt kontigban, anélkül, hogy a BAC szekvencia melyik darabjának megőrzését részesítették volna előnyben. A rések nélküli szekvenciáknak azonban nagyobb prioritása volt, mint a réseknek. Az összes elemet manuálisan ellenőriztük és szükség szerint beállítottuk a GPM 16 böngészővel. Amikor az összeszerelt Illumina-kontigumokat használtuk két BAC-alapú folytatás közötti rés kitöltésére, csak olyan Illumin-kontigumokat választottunk ki, amelyek teljesen összekapcsolhatták két szomszédos BAC-alapú folytonosságot, és ami fontos, ilyen átfedéseket ('minOverlapSeqToSeq = 1000 bp' a 'identitásSeqToSeq = 99% ") szükséges, hogy mindkét folytatás végén megjelenjen." Amikor ezekben a régiókban redundáns helyeket találtunk, a BAC-alapú szekvencia szekvenciákat mindig a végső genom-összeállításokban tároltuk.
Ez a dokumentum a ZS97 és MH63 indica rizsgenomok összeállításának kezdeti adatainak első kiadása, és a tudományos közösség két kiváló minőségű pszeudomolekulájának első változatát is tartalmazza. A DNS-szekvenálási technológiák és a szekvencia-összeállító programok gyorsan változnak, és az itt bemutatott adatsorok többféle szekvenálást tartalmaznak, amelyek felhasználhatók új módszerek és szoftvereszközök kifejlesztésére tesztbevitelként.
További részletek
Hogyan idézhetem ezt a cikket: Zhang, J. és mtsai. Indica rizs két referencia genomjának generálása PacBio hosszú leolvasási és Illumina szekvenálási adatokkal. Sci. 3. adat: 160076 doi: 10.1038/sdata.2016.76 (2016).
Adatok idézése
GenBank KG737749 - KG771717 (2013)
GenBank KG702200 - KG737748 (2013)
Archívum olvasásra NCBI SRP071597 (2016)
NCBI Sequence read archive SRP071598 (2016)
NCBI Sequence read archive SRP071944 (2016)
- Szerelmes Tomáš Klusba, két hét után tudta, hogy Tamarával szeretne kisbabát
- A biológiai sokféleség csökkenésének megállítása, valamint egészséges és fenntartható élelmiszer-rendszer létrehozása Európában
- Wellness a brnói Fontana szállodában félpanzióval két és 6 éves korig ingyenes gyermekkel, Csehország - Brno
- A Yelp a gépi oktatással készíti el a kedvenc ételek listáját
- A két gyilkosság közül négy lehet