elemeket
- DNS szekvenálás
- genetika
- genom
- A populációgenetika
Az óriáspanda genom a nagy emlősök genomjának első jelentett de novo összeállítása, amelyet a következő generációs szekvenálási módszerekkel értek el. Ez az értékelés tükrözi a genomszekvenálás egyre csökkenő költségei felé mutató tendenciát.
A Li és munkatársai e számának tanulmánya. A Pekingi Genomikai Intézet 1. oldala (311. oldal) két okból is figyelemre méltó - bemutatja az óriáspanda (Ailuropoda melanoleuca) genom elsődleges szerkezetét, és egy emlősökből összeállított, kiváló minőségű de novo genom első kiadványa. következő generációs szekvenáló (NGS) gépek segítségével. A pandát, amelynek genomját szekvenálták, Jingjingnek hívják; hozzávetőlegesen 3000 megmaradt lény egyike. Amellett, hogy Kína az egyik kínai nemzeti kincs, a panda érdekes helyet foglal el az evolúciós fában - emberek és kutyák között. Szokatlan bambusz étrendre is támaszkodik. Lenyűgöző tantárgyat nyújt a genom tanulmányozásához, amely kihasználhatja annak alapbiológiájának ismeretét, és megalapozhatja a pandapopuláció genetikáját.
Sokak számára azonban az lesz a legnagyobb érdeklődés, hogy az óriáspanda képviseli az NGS módszerekkel elért ilyen bonyolult genom első publikált de novo együttesét. Ezek a módszerek nagyon rövid sorrendű leolvasást eredményeznek, sokkal nagyobb mennyiségben és alacsonyabb költségek mellett, mint a hagyományos Sanger-technikák. Noha tíz vagy több NGS genomról számoltak be (a legutóbbi becslések szerint genomonként 10 000–50 000 USD), mindegyiket szekvenálással készítették és hasonlították össze a szekvenciákat egy emberi referenciával. Nem oldották meg egy teljesen új szerelvény olyan összetett problémáját, mint Li et al. 1 tett. Más csoportok, amelyek küzdöttek az NGS 3-gigabázos emlősök genomjának újszerű összeállításával, megvizsgálják a panda adatok módszereit és minőségét, és megkérdezik, hogyan tükrözhetik ezeket a tanulságokat más folyamatban lévő genomprojektek. A pénzügyi ügynökségek szintén figyelemmel kísérik ezt a fejlődést, a genomszekvenálás költségeinek folyamatos csökkenő tendenciáját várva.
Nem minden emlős genomiális szekvencia azonos minőségű: a 2-es emberi genom kezdeti kialakítását 2001-ben készítettük átfedő Sanger-szekvenálással, amelyet klasszikus nukleotidszál-terminációs módszerekkel 3 állítottunk elő. A "lényegében elkészült" 4-es szekvencia 2003-ra történő kifejlesztése, amely majdnem annyi erőfeszítésbe és pénzbe került, mint az eredeti terv, az emberi genom szekvenciájában a koherenciánál és pontosságánál csúcsosodott ki, mint a tervezés, és csak néhány hiányossággal. az egész 3 gigabázisban marad. Azóta csak az egér részesült előnyben a kész 5 genomból, és a többi publikált genomiális szekvencia, mint például a rhesus makákó 6, a 7-es kutya, a 8-as patkány és a 9-es tehén, kiváló minőségű fogalmak, amelyekre hat-nyolc -szeres genom lefedettséget Sanger szekvenálási módszerekkel értek el, speciális összeszerelő szoftverrel, amely kiszámította az átfedéseket.
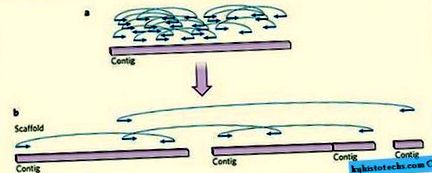
A tipikus Sanger-féle fogalmi szekvenciák 20-200 kilobázisú, úgynevezett összefüggő N50-ekkel rendelkeznek, amelyekben az összes bázis fele ekkora vagy annál hosszabb összefüggő szekvenciafragmensekben helyezkedik el. A megszakítás nélküli szekvencia ilyen hosszúsága lehetővé teszi a legtöbb gén egyedi összefüggő egységként történő tanulmányozását. A folytatásokat összeillesztve hosszabb szerkezetek vagy állványok képződnek, amelyek gyakran hosszú, többbázisúak (1. ábra). Általánosságban elmondható, hogy a magasabb állványértékek és az összefüggő N50 hasznosabb, mert lehetővé teszik a genomi tulajdonságok tanulmányozását a kontextusban - például a géneket összes exonjukkal (kódoló régiójukkal) sorrendben és a szomszédos szabályozó szekvenciákkal együtt. A kiváló minőségű szekvenálás megköveteli a hamis kapcsolatok megelőzését, amelyek a távoli szekvenciák helytelen összekapcsolásával felfújják az N50 kontig és állványt. Az a tény, hogy a korábbi projektekben használt Sanger-leolvasások hosszabbak és pontosabbak, mint az NGS-adatok (általában körülbelül 1000 bázis a kevesebb, mint 100 bázishoz képest, 0,1% vs.> 1% hiba), sokakban megkérdőjelezte, hogy költséghatékonyak-e., NGS módszerekkel kiváló minőségű tervező készletek állíthatók elő.
a, A genomot először sok apró töredékre osztják fel, amelyek végeit szekvenálják, így párpároknak nevezett szekvenciákat hoznak létre, amelyeket ismert távolság választ el egymástól (görbékkel összekötött kék nyilak). Ezeket az olvasmányokat olyan számítógépes algoritmusok segítségével állítják össze, amelyek egymással átfedő szekvenciákat egyesítenek, hogy összefüggő szekvenciafragmentumokat vagy folytatásokat képezzenek. b, A folytatások nagyobb állványokban vannak elrendezve, felhasználva a szekvencia hasonlóságára és a párok közötti távolságra vonatkozó információkat. Az ábrán egy állvány látható, amely szekvenciális kontigumokból áll, várható méretű szétszórt résekkel, a kontigókat összekötő párok alapján. A különböző szekvenálási technológiáknak eltérő a hibaaránya és az olvasási hossza, de az elvek ugyanazok. Egy tipikus Sanger merítési készlet körülbelül 1000 bázis hosszúságú adatokat generál, míg a Li és mtsai által alkalmazott következő generációs módszerek. Az óriáspanda genom-összeállítás 1-je sokkal kisebb (50–75 bázis hosszú).
Teljes méretű kép
Li és mtsai. 1, hogy ez a feladat egyszerűnek, ellenőrizhetőnek - sőt könnyűnek - tűnjön. A szerzők az Illumina szekvencia platform használatával figyelemre méltó 73-szoros teljes lefedettséget generáltak a panda genomra 50 és 75 bázisolvasással - ez egy tipikus összehasonlítható Sanger-féle genomprojekt átlagos lefedettségének körülbelül nyolcszorosa. A nagyobb lefedettségből adódó megnövekedett redundancia kompenzálhatja a kevésbé megbízható szekvenciaadatokat a végső konszenzus szekvencia pontosságának javítása érdekében. Ezenkívül a szerzők a legjobb adatok körülbelül kétharmadát használták fel egy összefüggő szekvencia létrehozásához, aminek eredményeként az N50 értéke 40 kilobázissal növekedett. Ezeket a folytatásokat kombinálva állványokat kaptunk N50-vel 1,3 megabázissal. Így a genom-összeállítás viszonylag hosszú szekvenciákat tartalmaz, de továbbra is fragmentálva marad 8080 állványban, szemben a kutyáknál kevesebb mint 100-zal. Az ilyen töredezettség meghiúsítja a genomi adatok használóit, és kizárja egyes funkciók elemzését, például a nagy szerkezeti variánsokat, de sok genomi elemzés még mindig lehetséges.
Li et al., Támaszkodva erre az új genomiális összeállításra. Az 1. ábrán a panda genomi szekvenciájának számos érdekes tulajdonságát találtuk, amelyek a panda biológiához kapcsolódnak. Bár az állatnak a húsevőkre jellemző génkészlete van, növényevő étrendje főleg bambuszból áll. Ezzel az étrenddel összhangban úgy tűnik, hogy az umami ízreceptort kódoló gén a fűszeres vagy húsos étel tulajdonságai miatt a pandában nem működik. Összességében a Jingjing genom magas szintű heterozigozitást mutatott (ahol a szekvencia különbözik az anyai és az apai kromoszómák között), amelyet a szerzők optimista módon úgy értelmeznek, hogy tükrözik a többi faj magas genetikai sokféleségét. Mivel csak egy állatot vizsgáltak, ezzel a kulcskérdéssel, miszerint az általános genetikai variabilitás a pandafajokban fordul elő, közvetlenül nem foglalkoztak. Nyilvánvaló, hogy sokkal több munkára van szükség, hogy biztosak legyünk ebben a következtetésben.
Az összeszerelési algoritmusokat bonyolító genom tulajdonságok közé tartoznak a gyakori, egyszerű DNS ismétlések, transzpozon inszerciók (mobil DNS), strukturális elrendezés és a közelmúltban eltérő szegmentális duplikációk. A szegmentális duplikációk különös kihívást jelentenek, mert egy hasonló szekvencia viszonylag hosszú szakaszait jelentik. Li és munkatársai azonban arról számolnak be, hogy a pandának viszonylag kevés van. Érdekes módon mind az egér 5, mind a 7 kutya genom együttese a szegmens duplikációjának nagyon alacsony arányát jelezte, de a későbbi munkák mindegyik esetben azt mutatták, hogy az egyes genomok 4, 5% -a duplikálódott így. A különbség a szerelvény "süllyedésének" eredménye volt, ahol az összeszerelési program akaratlanul is átfedésben volt hasonló szekvenciákkal. Ha ez megtörténhet az egér- és kutyaprojektekben használt viszonylag hosszú és pontos Sanger-adatokkal, akkor ez megtörténhetett a jelenlegi tanulmányban is.
A panda genom-szekvenálási projekt körülbelül 30 ciklusnak megfelelő Illumina-gépet igényelt, ami hozzávetőlegesen 900 000 USD nyers adatgyártásnak felel meg. Ez azonban legalább egy évvel ezelőtti adatgyártás sebessége, azóta csökkent a gép teljesítménye és az ennek megfelelő adategységenkénti költség. Az NGS módszerek jobban párhuzamosan működnek, mint a Sanger módszerei, és ciklusonként több millió olvasást tesznek lehetővé a Sanger több száz szekvenciájához képest. Mindezeket figyelembe véve, és még az adatmennyiség kezeléséhez szükséges számítási infrastruktúra, nagy memória-processzorok és tárolókapacitás miatt az óriáspanda-genom szekvenálásának költsége jóval alacsonyabb, mint a Sanger-féle genomszekvenálási projektek költségei, amelyek tízmillió dollár/genom. Ez a rendkívüli költségcsökkentés a pontossággal, koherenciával és teljességgel kapcsolatos bizonytalanságok ellenére azt jelenti, hogy Li és munkatársai 1 támogatni fogják az NGS módszerek alkalmazását más de novo genom-összeszerelési projektekben.
A panda-genom összeállítás teljességének és pontosságának teljes körű objektív értékelése megköveteli a projekt különböző módszerekkel történő megismétlését és a verziók közötti különbségek alapos vizsgálatát. Ez a közeljövőben nem fog megtörténni, de a nagylelkű nyilvánosságra hozatal jelenlegi egyezményével minden jelentés nyilvános nyilvántartás tárgyát képezi, és valószínűleg felül fogják vizsgálni, ahogy a technológia fejlődik, mások pedig ugyanazt a fajt vizsgálják. Mint minden genomadat, a pandakészlet túléli az időpróbát és a független minőségi teszteket.
Hozzászólások
Megjegyzés benyújtásával elfogadja az Általános Szerződési Feltételeinket és a közösségi irányelveket. Ha bármi sértőnek vagy összeegyeztethetetlennek tűnik a feltételeinkkel vagy irányelveinkkel, jelölje meg nem megfelelőként.
- Csokoládé desszert expressz étrend, 25,5 g - A természet egészséget hoz
- CK Limit Cities and of the West of USA - Amerikai Egyesült Államok - az Egyesült Államoktól nyugatra
- Biokémiai vérvizsgálati indikációk elnevezéshez és dekódoláshoz Megfelelő egészség az iLive-ban
- Atópiás ekcéma - kezelés-diéta - 6 hét - Fórum
- Hörghurut - táplálék-kiegészítők és STARLIFE-vitaminok