elemeket
absztrakt
Anyagok és metódusok
Etikai megállapítás
Az ebben a vizsgálatban leírt összes állatmunkát az Arkansasi Egyetem Állatgondozásával és -használatával foglalkozó intézményi bizottság (IACUC) hagyta jóvá, és minden kísérletet a jóváhagyott irányelveknek és előírásoknak megfelelően végeztek.
Állatokkal végzett kísérlet megtervezése
Vérgyűjtés és DNS-kivonás
A 14., 41. és 49. napon mikroorganizmus-elemzés céljából vérmintákat vettek 5 látszólag egészséges madárból tollban, a 240 vérminta megszerzésére (5 madár per toll × 16 toll mintavételi korban x 3 mintavételi korosztály). További 12 vérmintát vettünk BCO-n átesett madarakról (n = 12; 5 és 7 minta 41, illetve 49 napos kortól), ami lehetővé tette, hogy összesen 252 mintát elemezzünk 16S rRNS profilozás céljából. A vérmintákat aszeptikusan vettük a szárnyas vénából EDTA Vacutainers alkalmazásával. 1 ml vérmintát centrifugáltunk (5000 fordulat/perc 5 percig szobahőmérsékleten) mikrocentrifuga segítségével, és 200 μl buffy bevonatot összegyűjtöttünk egy steril lamináris áramlási kamrába, és -20 ° C-on tároltuk. A buffy bevonatokból genomi DNS-t extraháltunk BiOstic® Bacteremia DNS izoláló készlet (MoBio) felhasználásával a gyártó utasításainak megfelelően. A DNS-mintákat Qubit 2.0 fluorométerrel (Life Technologies) elemeztük mennyiségük és tisztaságuk szempontjából, és -20 ° C-on tároltuk.
PCR protokoll a 16S rRNS gén amplifikálásához
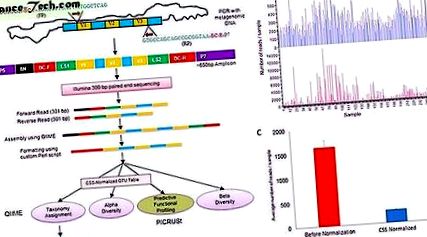
( a ) Folyamatábra, amely a könyvtár előkészítését, a QIIME elemzéshez elfogadható leolvasások formázását ábrázolja, amelyeket a LefSe és a PICRUSt alkalmazásával tovább elemeztek. * A PICRUST elemzéshez standardizált OTU táblázatot használtak. (FP = előreindító, RP = fordított primer, V = a 16s RNS gén variábilis régiója, P5 és P7 = Illumina szekvencia primerek, N = véletlenszerű nukleotid, BC-F = előreindító vonalkód, BC-R = fordított primer vonalkód) és LS = linker szekvencia). ( b ) Oszlopdiagram, amely 252 minta eloszlását mutatja az OTU táblázat előtt (piros) és normalizálás után (kék). c ) Oszlopdiagram, amely a mintánkénti átlagos leolvasások számát mutatja normál hibával (piros) és a normalizálás után (kék).
Teljes méretű kép
Adatelemzés
A 16S rRNS-szekvencia elemzését leíró folyamatábra a 3. ábrán látható. 1a. Az illumin szekvenálás párosított végének előre és hátra történő leolvasását a mikrobiális ökológia kvantitatív betekintése, a QIIME 1.9.1 25 segítségével állítottuk össze a join_paired_ends.py szkripten keresztül a fastq-join módszerrel. Ezután az előreolvasott (BC-F) és a visszaolvasó (BC-R) vonalkód-szekvenciákat 8 nukleotid véletlen szekvencia és 27F és 533R primer eltávolítása után összekötöttük és összekötöttük (5 'vég) az összeszerelt szekvenciával (3' vég). szekvenciák Perl saját szkriptjével, amely egy gyors q fájlt készített, amely kompatibilis volt a későbbi QIIME segítségével végzett elemzéssel.
Az olvasásokat az UCLUST 26 és az operatív taxonómiai egységek (OTU) segítségével csoportosítottuk, a referencia bezárásának lehetőségével a GreenGenes adatbázis 13_8 kiadásával. Az OTU BIOM táblázatot (biológiai megfigyelési mátrix) QIIME (normalize_table.py) alkalmazásával normalizáltuk a kumulatív összegösszeg (CSS) módszer alkalmazásával, amelyet azután taxonómiai hozzárendelésekhez, alfa sokféleség becsléséhez, biomarkerek azonosításához és a funkcionális metagenóm tartalom előrejelzéséhez használtunk. Az alfa diverzitás indexeket egyirányú varianciaanalízissel (ANOVA) hasonlítottuk össze, és a post hoc elemzést Tukey-Kramer HSD módszerrel hajtottuk végre. A metaadat-csoportok közötti hasonlóság-elemzést (ANOSIM) a QIIME-vel végzett súlyozatlan UniFrac távolságmérő metrikával végeztük el (Compare_categories.py). Ezenkívül master koordinációs elemzéseket (PCoA) végeztek a béta változatosság becsléséhez QIIME-val (24), súlyozott és súlyozatlan UniFrac mérőszámok alkalmazásával, különböző mintavételi mélységekben.
A biomarkerek azonosítását a lineáris diszkrimináns elemzés (LEfSe) 27 hatásának nagyságával végeztük. A csirke vér mikrobiomjainak metagén funkcionális tartalmát PICRUSt-mal (a közösségek filogenetikai kutatása nem megfigyelt állapotok rekonstrukciójával) jósoltuk 16S rRNS génszekvenciák alkalmazásával 28. A baktériumhálózatot a Cytoscape 3.2.1 alkalmazásával tettük láthatóvá.
az eredmény
A QIIME demultiplexelés és a minőségi szűrés 4, 153, 965 összeállított szekvenciát eredményezett 40 és 580 bp között, átlagos hossza 469 bp, ami kimutató szekvenciák jelenlétét jelzi aberráns PCR-amplifikáció vagy rossz illesztés miatt. Ebben a tanulmányban a zárt OTU referenciamódszert alkalmaztuk, mert egy új típusú mikrobiómát vizsgálunk, amelynek közösségi struktúrája, sokfélesége és tagsága nagyrészt ismeretlen. Összehasonlítottuk azonban a zárt referenciamódszer alkalmazásával kapott eredményeket a nyílt referencia módszerekkel és a de novo OTU szedési módszerekkel is (2. kiegészítő táblázat). Bár a nyílt referencia és a de novo OTU választási módszer magasabb OTU-khoz (átlagosan 196, illetve 218), valamint a minta magasabb értékéhez vezetett, jelentős arányokat rögzítettek (átlagosan 72, 5 és 79, illetve%). ), amelyek a jelenlegi GreenGenes adatbázis 13_8 egyik taxonómiai csoportjába sem tartoztak. Amellett, hogy a későbbi elemzés során hatékonyan eltávolítottuk az összes zajt vagy kiméra szekvenciát, a zárt referencia OTU gyűjteményt részesítettük előnyben a másik két módszerrel szemben.
A zárt OTU referenciatábla átlagos mintamélysége 1621 volt, minden mintára (± 139, 07), amint az a 3. ábrán látható. Az OTU táblázatot QIIME-val végzett kumulatív skálázás (CSS) alkalmazásával normalizáltuk (1b. Ábra, c). Noha a minták átlagértékei több mint 6-szoros normalizálással csökkentek (mintánként 266, 55), csökkentették a standard hibát is (± 7, 01). A CSS normalizált OTU táblázatot használtuk a taxonómia, az alfa diverzitás becslések, az LEfSe elemzés és a PICRUt elemzés hozzárendeléséhez. A béta változatosság elemzését különböző mintavételi mélységekben és ANOSIM-ban végeztük 999 permutációval, a súlyozatlan UniFrac mérőszám segítségével, ugyanazon mintavételi mélység mellett, 400 képkockánként.
Rendszertan hozzárendelése
a ) Oszlopdiagram törzsszinten átmérővel Az egyes oszlopok felett található különböző betűk jelentős eltéréseket jeleznek (ANOVA, Tukey-Kramer teszt, 4. o.) .
Dobozkerítés kísérleti kategória távirányítóval. A mezők a középső, a 25. és a 75. percentiliseket mutatják, az értékek az alsó és felső szélső értékeken kívül esnek. A különböző betűkkel ellátott dobozok jelentősen különböznek (p
a ) Egészséges (n = 171) és BCO (n = 9) csirkék PCoA-diagramja súlyozott UniFrac mérőszámok alkalmazásával, ugyanazon mintavételi mélység mellett, 400 olvasás mintánként. ( b ) 14 napos (n = 30), 41 napos (n = 41) és 49 napos (n = 36) régi csirkék PCoA-diagramja súlyozott UniFrac mérőszámokkal, mintánként ugyanazon mintavételi mélységben, 1000 képkocka mellett. ( c ) A kezdő (n = 67) és a befejező (n = 154) csirkék etetett étrendjeinek PCoA diagramja súlyozatlan UniFrac mérőszám alkalmazásával, mintánként 200 képkocka/minta azonos mélységben.
Teljes méretű kép
Az egészséges és a BCO csirkék baktériumközösségeiben mutatkozó szignifikáns különbséget hierarchikus csoportosítás is szemléltette. A BCO csirkéket 49 napos csirkékkel különítettük el az egészséges csirkéktől. A 41 napos BCO csirkék azonban nem mutattak szignifikáns csoportosulást (5a. Ábra). A csirkék (252 minta) és az OTU (baktériumfajok) közötti hálózati elemzés bizonyos fokú felbontást mutatott a BCO és az egészséges csirkék közötti interakciós modellekben (5b. Ábra), ami különböző baktériumközösségekre utal.
a ) Egészséges és BCO csirkék hierarchikus csoportosítása mikrobiális vér alapján. A filogenetikai fát a FigTree V1.3.1 szoftver segítségével állítottuk elő, súlyozott UniFrac mérőszámokkal, előre normalizálási adatok felhasználásával. A zöld és a piros vonal a BCO csirkéket ábrázolja 41 és 49 napig. A fekete vonalak egészséges csirkék. A sor végén lévő szám a csirkeminta azonosítóját mutatja. b ) A Cytoscape V3.2.1 alkalmazásával előállított csirkevér mikroorganizmusok baktériumhálózata. A vörös, kék és fehér csomók a BCO csirkéket, az egészséges csirkéket és az OTU-t képviselik. A zöld határ a BCO csirkehálózat, a narancssárga perem pedig egészséges csirkékben található.
Teljes méretű kép
Megállapítottuk, hogy az összes BCO madár csak két korcsoportból (41. és 49. nap) és két csoportból (W35-56 és W1-56) származott. Ezért elvégeztünk egy további béta-változatosság-elemzést, amelyhez az ezen korcsoportokhoz és az ajak-alapú csoportokhoz tartozó 77 minta részhalmazát használtuk, köztük 65 egészséges madarat és 12 BCO madarat. Az UniFrac súlyozott metrikákon alapuló ANOSIM elemzés nagyobb különbséget mutatott a BCO és az egészséges csirkék között (R = 0,5293, p = 0,001) a teljes adathalmazhoz képest (n = 252). Ábrán látható PCoA grafikon. A 6. ábra szintén támogatja az ANOSIM elemzés eredményét.
Egészséges (n = 51) és BCO (n = 9) csirkék PCoA diagramja súlyozott UniFrac mérőszámok alkalmazásával, mintánként 400 olvasásmintán ugyanazon mintavételi mélység mellett.
Teljes méretű kép
BCO biomarkerek
Az egészséges és a BCO csirkék között bőségesen eltérő taxonómiai csoportokat azonosítottunk a lineáris diszkrimináns elemzés (LEfSe) hatásának nagyságával, = 0,05, LDA pontszám legalább 2, relatív száma pedig 0,1 felett. Összesen 26 funkció szignifikánsan eltérő mennyiség az egészséges és a BCO csirkék között. Nemzetségi szinten a csirkék BCO vérmikrodiódái differenciálva voltak Staphylococcus, Granulicatella és Microbacterium, míg az egészséges csirkék Pseudomonas, Enhydrobacter és Aquabacterium dúsítottak (7b. Ábra). Azt is megállapítottuk, hogy a Firmicutes törzs BCO csirkékkel gazdagodott. Hasonlóképpen, az Alphaproteobacteriumok nagyon gazdagak voltak a BCO csirkékben, míg a Betaproteobacteriumok és a Gammaproteobacteriumok egészséges csirkéknél osztály szinten.
a ) Rendszertani cladogram készült LEfSe elemzésből. A vörös és a zöld BCO-val dúsított taxonokat és egészséges csirkéket tartalmaz. A fényerő arányos a taxon mennyiségével. ( b ) A BCO csirkékkel dúsított adókat piros színnel mutatják, negatív LDA pontszámmal, és egészséges csirkéket zöld állapotban, pozitív LDA pontszámmal (mindkét esetben> 3, 5). A két aláhúzás közötti taxont javasoljuk a GreenGene adatbázis neveként.
Teljes méretű kép
A csirkevér mikroorganizmusainak feltételezett funkcionális genetikai kapacitása
( a ) A KEGG COG-szintek relatív száma 1. szinten a csirkevér mikrobiómáiban. ( b ) A KEGG útvonal relatív bősége (≥ 0, 5) a JMP szoftver által létrehozott funkcionális útvonalak 3. szintjén. ( c ) differenciált karakterek (KEGG COG kategória, relatív bőség ≥ 0, 5), amelyeket LEfSe alkalmazásával állítottak elő, LDA pontszám ≥ 2, 8.
Teljes méretű kép
vita
Ez az első átfogó tanulmány, amely elemzi a nem emberi gerincesek vérében található baktériumos mikrobákat. Az elmúlt években egyre nagyobb az érdeklődés a test különböző szöveteihez kapcsolódó mikrobionok jellemzése iránt, különböző egészségügyi körülmények között. A sterilnek tekintett testrészeknek, mint például a vér 30, a gyomor 31, a hólyag 32, a tüdő 33, a csontok, a 4 ízületek és a 34 mellnek megvan az eredeti mikroorganizmusa. A szöveti mikrobiosis dysbiosis számos betegséggel társult, beleértve a szív- és érrendszeri betegségeket 10, a cukorbetegséget 35, az alkoholmentes zsírmáj betegségeket (NASHD) 36, a gyulladásos bélbetegségeket (IBD) 37, a pikkelysömör 38-at, az elhízást 39, a gyermekkori asztmát 39, funkcionális bélbetegség 40 és vastagbélrák 41. Itt megvizsgáltuk a csirkevér mikrobiózisát, hogy azonosítsuk a BCO-val összefüggő potenciális baktériumos biomarkereket.
A tanulmány adatelemzésének eredménye azt mutatta, hogy a brojlercsirkék vérében 30–40 OTU-ból álló baktériumközösségek léteznek, életkortól és egyéb környezeti vagy gazdaállapotoktól függetlenül. A béta-változatosság elemzése (4a. És 6. ábra), hierarchikus klaszteranalízis (5a. Ábra) és baktériumháló-elemzés (5b. Ábra), mikrobiális véren alapulva, és az előrejelzett vérmikrobionos metagenómán alapuló hierarchikus klaszterezés (8b. Ábra) arra utalnak, hogy a baktériumok A BCO vérében a madarak különböznek az egészséges madarak közösségeitől, ami bizonyos szelektív nyomások jelenlétére utal, amelyek hozzájárulnak a vér mikrobiomereinek eltolódásához a BCO madarakban. Azonban csak a teljes PD fa mutatott szignifikáns különbséget az alfa változatosságban a BCO-k és az egészséges madarak között, összhangban a filogenetikailag távoli vér mikrobiózissal a BCO madarakban, amint azt a béta változatosság elemzése mutatja (4a. És 6. ábra).
Publikálatlan kísérő vizsgálatunk során ugyanabból a nyájból származó vérmintákat közvetlenül oltottak be gazdag agar táptalajra. Az eredmény azt mutatta, hogy a telepek száma a 49. napon következetesen magasabb volt a huzalpadlókon (L35W és W56), mint az ágyneműkben (L56), ami arra utal, hogy a huzalpadló feszültsége valószínűleg elősegítette a bakterémiát a bélhámon keresztüli baktérium transzlokáció révén. Ezenkívül a telepek száma a 49. napon szintén folyamatosan magasabb volt a béna madaraknál, mint az egészséges madaraknál. Ezek az eredmények erős korrelációt jeleznek a huzal padlófeszültsége, a bakteriémia súlyossága és a BCO sántasága között.
A tanulmány egyik gyakorlati alkalmazása olyan baktériumos biomarkerek azonosítása volt, amelyek felhasználhatók olyan egyedi brojlercsirkék azonosítására egy korábbi életkorban, amelyek hajlamosak a későbbi életkorban a BCO kialakulására. Valójában a vizsgálat adatainak elemzése különböző szintű taxonómiai csoportokat azonosított, amelyek jelentősen gazdagabbak a BCO-madarakban, mint az egészséges madarak. Érdekes módon a Staphylococcus nemzetség egyike a 18 tulajdonságnak (köztük 3 nemzetségnek), amelyek jelentősen gazdagodtak a BCO mintákban, kiemelve ennek a nemzetségnek a fontosságát, amelyet gyakran izoláltak a BCO 3, 4, 5 elváltozásokból. Nemrégiben Al-Rubaye és mtsai. 44 kijelentette, hogy az S. agnetis nevű brojlerek kiváltása, amely a tanulmányában a BCO elváltozásokból leggyakrabban izolált staphyloccosus faj volt, jelentősen megnövelte a sérülést 10-ről (kontroll) 40% -ra, míg egy másik Enterococcus faecalis izolátummal való fertőzés csökkentette a izom-csontrendszeri rendellenességek előfordulása, Ez jelezheti az S. agnetis mint a BCO kórokozójának potenciális fontosságát a brojlercsirkéknél, bár a 16S rRNS génprofilozásra vonatkozó adatunk a korlátozott taxonómiai hozzárendelés miatt nem nyújtott érdemi információt a Staphylococcus fajokról.
Ebben a tanulmányban azonban a BCO-t csak a 41 és 49 napos madaraknál mutatták ki, és ezért a baktériumok biomarkereinek korlátozott értéke van a BCO-fogékony madarak korai diagnózisában. Ezt a szempontot gondosan figyelembe kell venni a jövőbeni vizsgálatok kísérleti tervezése során, hogy lehetővé tegyék a potenciális bakteriális biomarkerek azonosítását fiatal madár vérmintákban, amelyek előre jelzik a BCO fejlődését idősebb korcsoportokban.
Az ebben a vizsgálatban elemzett vérmikrobák jelentős hatással vannak a brojlercsirkék egészségi állapotára, beleértve a BCO patogenezisét, amint ezt a tanulmány kimutatta, valamint a brojlercsirkék egyéb betegségeit vagy stresszállapotait.
További részletek
Hogyan lehet idézni ezt a cikket: Mandal, RK et al. A mikrobiális vér és annak potenciális összefüggésének vizsgálata brojlereknél bekövetkező osteomyelitis (BCO) baktériumok chondronecrosisával. Sci. ismétlés. 6., 25882; doi: 10.1038/srep25882 (2016).